関数をコマンドラインツールとして実行するpythonライブラリcommandrのご紹介
commandrのご紹介
最近みつけた便利なpythonライブラリ "commandr"というライブラリがあります。インストールはpip install commandrで入ります。このライブラリは、適当につくったpythonの関数に、@commandでデコレートすると、定義した関数がコマンドラインとして実行できる、という大変便利なものとなっております。tellapart/commandr · GitHub に詳しい使い方がのっていますが、基本は非常にシンプルなデコレータです。
使い方あれこれ
from commandr import command, Run @command def say(name=""): """ Arguments: name - your name """ print "Hi! {}".format(name) if __name__ == "__main__": Run()
上記のファイルをcmd.pyと保存して、python cmd.pyを実行してみます。sayは引数として受け取った文字列を表示するだけの関数です。
A command must be specified. General Commands: help - Prints the global help message listing all commands. say -
sayというコマンドが追加されていることがわかります。
python cmd.py say --name yukkeと実行すると、
Hi! yukke
pythno cmd.py say --helpは関数に書いたdocstringを表示してくれます。
Documentation for command 'say':
----------------------------------------
Arguments:
name - your name
----------------------------------------
Usage: hoge.py say [options]
Options without default values MUST be specified
Use: hoge.py help [command]
to see other commands available.
Options:
-h, --help
-n NAME, --name=NAME [default: ""]
docstringも見やすいですね。また、@command(category="analysis")などと書くと、コマンド群をカテゴリに分類しておけます。
なぜ便利か?どんなときに使えるか?
commandrは、データ解析をしている人には大変便利なツールとなり得ます。あるプロジェクトや仕事で、込み入った解析をするとなると、自作のクラスや関数を沢山書いて、それをライブラリとしてロードして、分析・解析をすることになります。このとき、pythonの標準的なディレクトリ構成は、
App/src/{プロジェクトのライブラリ}
App/scripts/{分析を実行するコード}
App/tests/{ライブラリのテストコード}
となっているかと思います。僕の場合ですが、自作のライブラリは、src以下にあり、それをscripts以下に設置した分析用のコードから呼び出す、という構成を取っています。
ところが、パーサーやアルゴリズムの実装に比べて、解析用のコードはtry-and-errorで進めていくものなので、開発中のコードは特に、実行コマンドは同じでも出力が異なることは日常茶飯事です。シェルのヒストリに残った昨日の./scripts/run.pyと今日のそれは、出力が同じであることのほうが稀です。
こういった状況において、入出力ファイルの名前やアルゴリズムのパラメータなどを出来る限りパラメータ化し、コマンドライン引数として実行することで、シェルのヒストリに記録しておく、というのは非常に有用なアプローチだと考えています。紹介したcommandrは、まさにそれを助けるためのライブラリで、研究活動における分析を出来る限り関数として定義し(冗長な名前でもよい)、@commandでデコレートすれば、コマンドラインとして実行可能となり、実行結果はシェルのヒストリに残るため、分析の再現性や分析のログとなるでしょう。
pythonでコマンドラインツールを書くことはさほど難しくはありませんが、やはり引数の処理などはめんどくさく、分析においてはあまり本質的ではありません。commandrは、pythonコードとdocstringからコマンドラインツールのインターフェースを自動生成しますので、あとから見返したときにも、実行方法が分からないというような悩みもある程度軽減されるはずです。ぜひお試しください。
/usr/bin/lessでもファイルをカラーでprettyにprintしたい
問題
- pagerならlvでもlessでもなんでもよいが、ソースコードや構造化されたファイル形式 (XMLやCSVなど)は、白黒よりもattributeやvalue、stringかintによって色が代わって表示されると中身を把握しやすい。
- しかしながら、標準のlessやlvにさまざまな形式に対応したシンタックスハイライト機能はない。
どうするか
- pygmentize (http://pygments.org/)というソフトウェアがあるのでこれを使うと、いい感じにシンタックスハイライトしてくれる。
pip install pygmentizeとpip install pygments-style-solarizedしてsolarized themeも入れておく。- こんな感じにxmlがpretty printされる。見やすい。

さらにひと味
sshfsでMacにリモートサーバをマウントする
問題
普段の研究は、計算資源の問題もあり(お手元のiMacではとても計算できない)sshで接続したunix計算サーバ上で作業することが殆ど。コーディングをしたり、データ解析をする分にはまったくCUIの世界で完結しているので問題はないのだけれど、解析結果を可視化することの必要性はやはりある。
むろん、Rやpythonでデータにかんするグラフを描くなどして可視化をすることはできるが、X11だと描画はきれいじゃないし、遅延したりあまり安定しない問題もある。ゲノム解析の分野では、DNAやRNAの塩基配列データをゲノム上で可視化する必要があり、またインタラクティブに座標の切り替えや拡大と縮小を必要とする。こうった操作はCUIでは難しく、GUIが得意とするところである。加えて、可視化するためだけに、わざわざデータを落とすのもめんどくさい。サーバでplotしてscpして確認して、サーバでplotしてscp、、、。
ということを踏まえると、CUIのリモートで生成されるデータとGUIのMacとの間をシームレスに行き来できる仕組みがあればよい。
解決策
sshfsを使うと、任意のディレクトリにssh先のサーバのディスクをマウントすることができる。
brew install sshfsするか、FUSE for OS X (http://osxfuse.github.io/)というのを入れる。mkdir /Volumes/remote# ディレクトリがないとbad mount pointとか怒られる。sshfs -o reconnect -p 22 soh.i@remote.01.jp:/home/yukke /Volumes/remote

これで、Finder上でリモートサーバのファイルを全てあたかもMacのファイルのように透過的に扱うことができるようになる。ここでコードを編集したりすることももちろん可能だ。マウントの解除は、umountすればいい。
やっぱり問題が
- まずFinderにmountできるのは大変便利だが、どうも通信速度の問題なのかファイルの取得や更新に時間がかかる。ずっとロード状態になってしまう。
- 仕組みは便利だけどあんまり使い物にならない。
結局
go-lang + g-languageでスクレイピングしてみた
A Tour of Goを途中でやったはいいものの、入門したっきりでgoを結局全然勉強してなかったので、とりあえず簡単なスクレイピングをしてみようと思って書いた。g-links (http://www.g-language.org/wiki/glinks)というタンパク質や遺伝子に関連する多くの情報を取得できるウェブサービスがあるので、今回はこれを題材に、実践的にコードを書きながら勉強してみた。
という感じになった。FOXP2_HUMANとQ9UPN9という二つのタンパク質の名前と、それに付随するGene ontologyのデータを出力している。
実行すると、
# AltName FOXP2_HUMAN CAG repeat protein 44 # AltName FOXP2_HUMAN Trinucleotide repeat-containing gene 10 protein # GOslim_component GO:0005737 cytoplasm # GOslim_component GO:0043234 protein complex # GOslim_function GO:0001071 nucleic acid binding transcription factor activity # GOslim_function GO:0003674 molecular_function # GOslim_function GO:0003677 DNA binding # GOslim_function GO:0008134 transcription factor binding # GOslim_function GO:0043167 ion binding # GOslim_process GO:0008150 biological_process # GOslim_process GO:0009790 embryo development # GOslim_process GO:0040007 growth # GOslim_process GO:0048856 anatomical structure development # GOslim_process GO:0050877 neurological system process --- # AltName TRI33_HUMAN Ectodermin homolog # AltName TRI33_HUMAN RET-fused gene 7 protein # AltName TRI33_HUMAN Transcription intermediary factor 1-gamma # AltName TRI33_HUMAN Tripartite motif-containing protein 33 # GOslim_component GO:0005654 nucleoplasm # GOslim_function GO:0003677 DNA binding # GOslim_function GO:0016874 ligase activity # GOslim_function GO:0043167 ion binding # GOslim_process GO:0007165 signal transduction # GOslim_process GO:0008150 biological_process # GOslim_process GO:0009058 biosynthetic process # GOslim_process GO:0034641 cellular nitrogen compound metabolic process ---
のようにスクレイピングした結果が得られる。
今日、初めてまともな機能をもつgoのコードを書いてみて感じたことは、
- 引数や返り値の型をちゃんと意識しないといけなくて、例えば引数が
[]byteなのか[]stringで戸惑う - なんかコンパイル後のバイナリのサイズがでかい
- 自分で定義した関数でbetterなerrorの返し方が分からない
- 沢山かいて慣れろ
ということくらいか。
まとめると、HTTPアクセスの仕方、正規表現の書き方、簡単な文字列の操作が分かってきた。今度は、methodとかinterfaceを勉強する。
季節感のあるプロンプトを出す
目的
一日の殆どをターミナルで過ごすわれわれにとっては、便利なカスタマイズをいろいろする部分だと思う。シェルの設定や補完や色とか。今回は、プロンプトの話。季節感のあるプロンプトを出したいと思った。
季節とは
- 冬なので、♨温泉♨だと思う。
やり方
.zshrcとかに以下の様な感じで書く。
FACE="Aichan" PROMPT="%F{red}♨ %f%F{magenta}%B$FACE%b %F{red}♨ %f%F{green}>> %f"

かわいい。
Unixで特定のプロセスだけを監視する
目的
- 特定のプロセスだけの状態を確認したかった。全部とか、特定のユーザーだけなら、
htopやtopコマンドでみるのがインタラクティブでよい。ただ、これでは特定のプロセスだけに絞り込んでみれない(出来るなら教えてください......)。
やり方
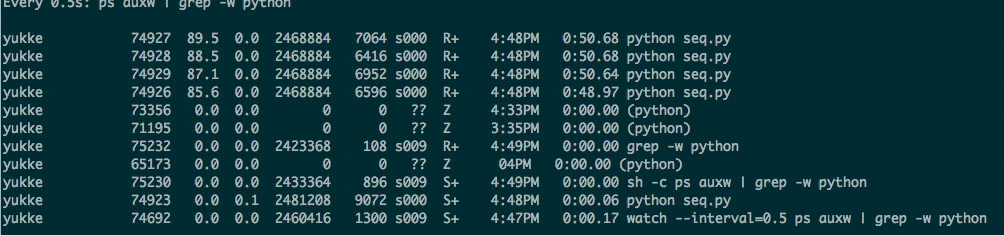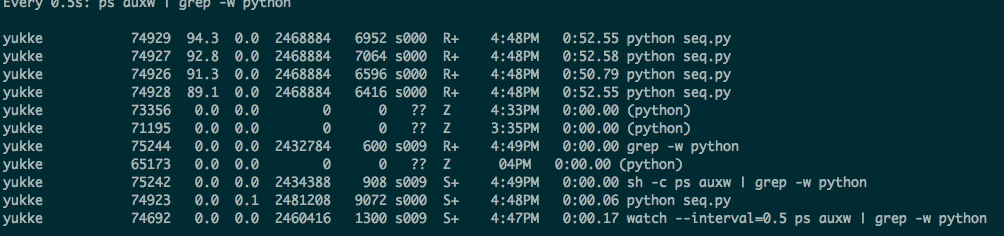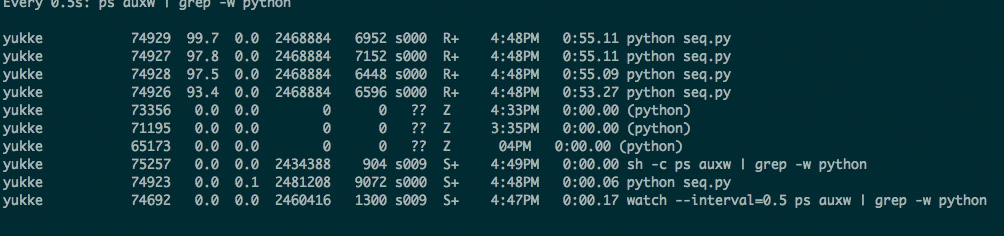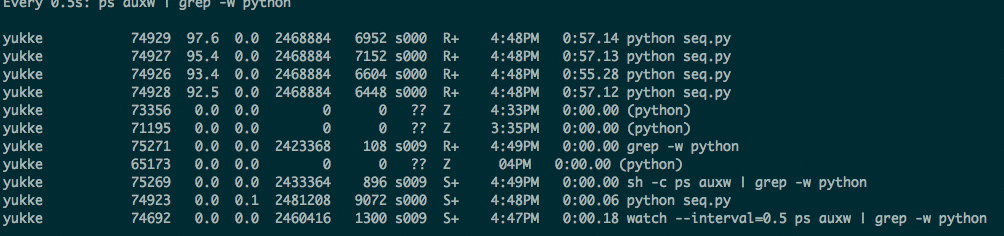
watch --interval=0.5 ps auxw | grep -w ${process_name}
ってやると、process_nameだけの状態が0.5秒おきに更新されてわかる。watch --interval=0.5 ps auxw | grep -w pythonとかやれば、pythonだけがわかる。便利。
watchコマンドは、ファイルとかの更新を自動で検知してくれて表示してくれるやつ。Macにはもともと入ってなくて、brew install watchした。
結果
以下がwatchコマンドの様子。並列処理を書いていて、立ち上げたwokerプロセスがちゃんとCPUを使っているかどうかとか、そういうのの確認をしたくて調べていた。
便利なので、ターミナルに一つウィンドウを立ち上て、これだけ表示して使っている。